Aktywne Wpisy

KosmicznyPaczek +66

Rodrigo_Borgia +77
Moje prawo jazdy obchodzi dzisiaj trzydzieste urodziny. Nie dzwońcie po śmieciarkę bo nie mam na nią uprawnień
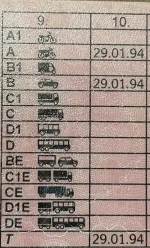
źródło: IMG_2381.jpg
PobierzSkopiuj link
Skopiuj link


źródło: IMG_2381.jpg
PobierzWykop.pl
W rdkicie można "robić" reakcje chemiczne określając substraty, produkt(y?). Można numerować atomy - generanie wszystko co SMARTS przewiduje. Reakcja z dwoma substratami ("." rozdziela substraty a po ">>" jest produkt):
r3=AllChem.ReactionFromSmarts('[C:1]=[C:2].[C:3]=[*:4][*:5]=[C:6]>>[C:1]1[C:2][C:3][*:4]=[*:5][C:6]1')
Potem uruchomienie RunReactants z krotką substratów powinno zwrócić listę(albo krotkę) z możliwymi produktami.
przykład z dokumentacji (bloga). Jednak nie działa:
p2=r3.RunReactants((Chem.MolFromSmiles('OC=C'), Chem.MolFromSmiles('C=CC(N)=C')))
daje błąd:
TypeError: No topython (by-value) converter found for C++ type: boost::sharedptr
Co jest winą biblioteki Boost w wersji 1.60. W starszych działa. Problem znany i jest patch https://github.com/boostorg/python/pull/59. Nie znalazłem informacji czy 1.61 ma już to poprawione.
W archu jest 1.60-5. Też brak info czy poprawili tam ten błąd.
W archu jest w aur rdkit ale wciąż stary, tak samo z boostem (ale tutaj od wydania nowej wersji minęło ~ 2 tyg więc może się ogarną).
Tym sposobem zamiast nauki reakcji w RDKicie będzie zabawa z bibliotekami i wersjami :/
Btw. ciekawa zabawka ;)